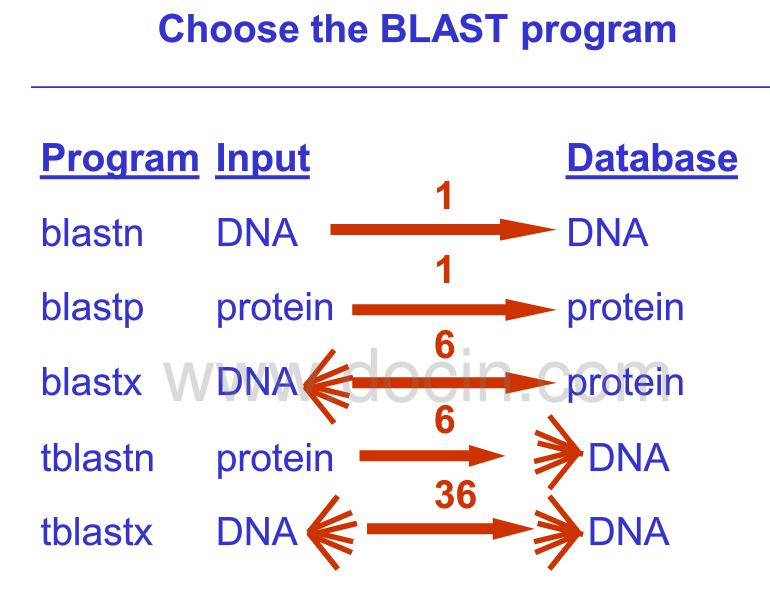
 NCBI Blastp 是什么意思?
NCBI Blastp 是什么意思?
770x613 - 70KB - PNG
 HaMStR: Profile hidden markov model
HaMStR: Profile hidden markov model
600x400 - 67KB - JPEG
 使用BLASTP搜索得到的结果,下面黑色的
使用BLASTP搜索得到的结果,下面黑色的
500x299 - 23KB - PNG
 同源建模方法整理
同源建模方法整理
913x500 - 41KB - PNG
 Identification of mammalian orthologs using
Identification of mammalian orthologs using
600x210 - 28KB - JPEG
 Genome plasticity and systems evolution in
Genome plasticity and systems evolution in
685x454 - 35KB - JPEG
 Modular organization and reticulate
Modular organization and reticulate
685x807 - 81KB - JPEG
 Identification of drought-responsive genes
Identification of drought-responsive genes
600x402 - 33KB - JPEG
 Reannotation and extended community
Reannotation and extended community
685x392 - 31KB - JPEG
 The predicted secretome and transmem
The predicted secretome and transmem
1200x681 - 70KB - JPEG
 The high-quality draft genome of peach (
The high-quality draft genome of peach (
685x493 - 37KB - JPEG
 分析混合微生物群落内细菌协同代谢关系,
分析混合微生物群落内细菌协同代谢关系,
640x410 - 29KB - JPEG
 新冠肺炎潜在特效药--弗林蛋白酶抑制剂
新冠肺炎潜在特效药--弗林蛋白酶抑制剂
880x605 - 74KB - JPEG
 Dramatic down-regulation of oxidoreductase
Dramatic down-regulation of oxidoreductase
1200x1553 - 148KB - JPEG
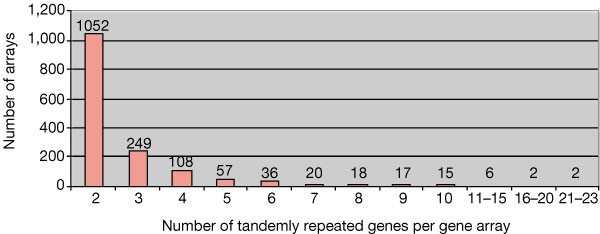 Analysis of the genome sequence of the
Analysis of the genome sequence of the
600x234 - 19KB - JPEG